Naukowcy z Wydziału Chemii UW, pod kierunkiem prof. Sławomira Filipka, uczestniczyli w powstaniu serwisu GPCRmd. Na platformie umieszczane są symulacje białek przydatne m.in. przy tworzeniu nowych leków. Od niedawna na portalu gromadzone są także symulacje białek wirusa SARS-COV-2. Publikacja na temat GPCRmd ukazała się w Nature Methods.
GPCRmd zawiera zestaw narzędzi analitycznych, który pozwala naukowcom z różnych dyscyplin wizualizować, analizować i przede wszystkim udostępniać symulacje dynamiki molekularnej układów biologicznych na poziomie atomowym. Analiza i wizualizacja tych symulacji wymaga wydajnych zasobów pamięci masowej i specjalistycznego oprogramowania.
Współpraca międzynarodowa
Prace nad portalem rozpoczęły się w 2017 roku. Strona ze wszystkimi funkcjonalnościami działa od końca 2019 roku. W jej tworzenie zaangażowani byli naukowcy współpracujący w międzynarodowym projekcie GLISTEN. Ich celem było stworzenie otwartej, interaktywnej i ustandaryzowanej bazy danych symulacji biologicznych. Wiodącą rolę w projekcie odegrał zespół prof. Jany Selent z Uniwersytetu Pompeu Fabry. W tworzeniu portalu uczestniczył też zespół prof. Sławomira Filipka z UW oraz naukowcy z Włoch, Grecji, Niemiec, Danii, Szwajcarii, Szwecji, Wielkiej Brytanii i USA.
Protokół używany do symulacji białek został wypracowany we współpracy z wszystkimi zaangażowanymi grupami badawczymi, jest publicznie dostępny, po to, aby zapewnić odtwarzalność wyników w różnych laboratoriach.
Dla kogo?
GPCRmd to portal dla biofizyków, krystalografów, biologów ewolucyjnych, specjalistów z chemii medycznej, ekspertów projektowania leków oraz inżynierii białek, bioinformatyków, a także studentów i nauczycieli oraz recenzentów i wydawców.
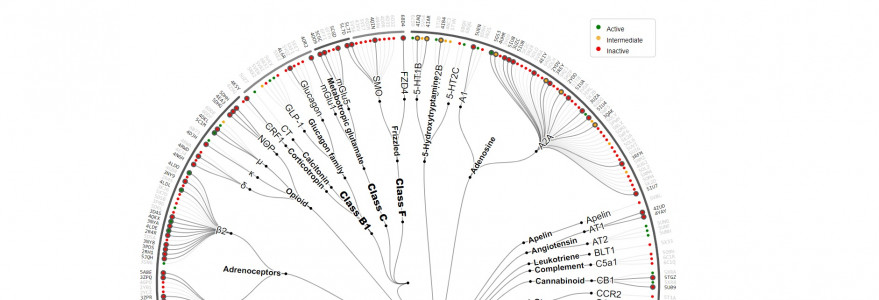
– Zawiera wyniki pełnoatomowych symulacji receptorów GPCR (receptorów sprzężonych z białkami G), które są niezbędne do sygnalizacji komórkowej w tym również w neuronach oraz ich wszechstronnej analizy za pomocą intuicyjnych narzędzi online – tłumaczy prof. Sławomir Filipek.
Od niedawna na portalu GPCRmd gromadzone są także symulacje białek wirusa SARS-COV-2, aby umożliwić różnym naukowcom przeprowadzenie badań podstawowych, jak i aplikacyjnych np. dotyczących projektowania leków.
– Dzięki portalowi naukowcy mają możliwość dokonywania samodzielnych analiz trajektorii receptorów lub ich zmodyfikowanych struktur, dowiedzieć się w jaki sposób przebiegają zmiany w receptorze w czasie symulacji, jakie jest oddziaływanie z ligandami (małymi cząsteczkami np. lekami), lipidami i innymi białkami – mówi profesor.